EvolutionaryScale zeigt KI, die Hunderte Millionen Jahre Proteinevolution in Sekunden simuliert

Ein neues KI-Modell namens ESM3 kann durch Training auf von der Evolution erzeugten Daten funktionale Proteine generieren, die in der Natur Hunderte Millionen Jahre bräuchten, um sich zu entwickeln.
Wissenschaftler des Unternehmens EvolutionaryScale haben ein großes KI-Modell namens ESM3 entwickelt, das in der Lage ist, die Evolution von Proteinen zu simulieren und dabei neue funktionelle Proteine zu erzeugen. In einer Demonstration dieser Fähigkeit erzeugte das Modell ein grün fluoreszierendes Protein (GFP) namens esmGFP, das nur 58 % Sequenzähnlichkeit mit dem nächstverwandten bekannten fluoreszierenden Protein aufweist.
Das Modell wurde auf 2,78 Milliarden natürlichen Proteinsequenzen, 236 Millionen Proteinstrukturen und 539 Millionen Proteinen mit Funktionsannotationen trainiert. Insgesamt hat ESM3 während des Trainings 771 Milliarden Token verarbeitet.
Video: EvolutionaryScale
ESM3 verarbeitet dreidimensionale Strukturen von Proteinen besser als ältere Modelle
Im Gegensatz zu herkömmlichen Sprachmodellen, die nur auf Textdaten trainiert werden, lernt ESM3 aus diskreten Token, die die Sequenz, die dreidimensionale Struktur und die biologische Funktion von Proteinen repräsentieren. "Proteine können als in einem organisierten Raum existierend betrachtet werden, in dem jedes Protein von jedem anderen Protein, das sich durch ein einziges Mutationsereignis unterscheidet, benachbart ist", erklären die Forscher.
Das KI-Modell ESM3 lernte, diesen Raum zu verstehen, indem es Vorhersagen über die nächsten Token in Proteinsequenzen traf - ähnlich wie Sprachmodelle Wörter in Sätzen vorhersagen. Im Gegensatz zu reinen Sprachmodellen hat ESM3 jedoch eine neuartige Architektur, die "geometrische Aufmerksamkeit" nutzt, um die dreidimensionale Struktur von Proteinen effizient zu verarbeiten.
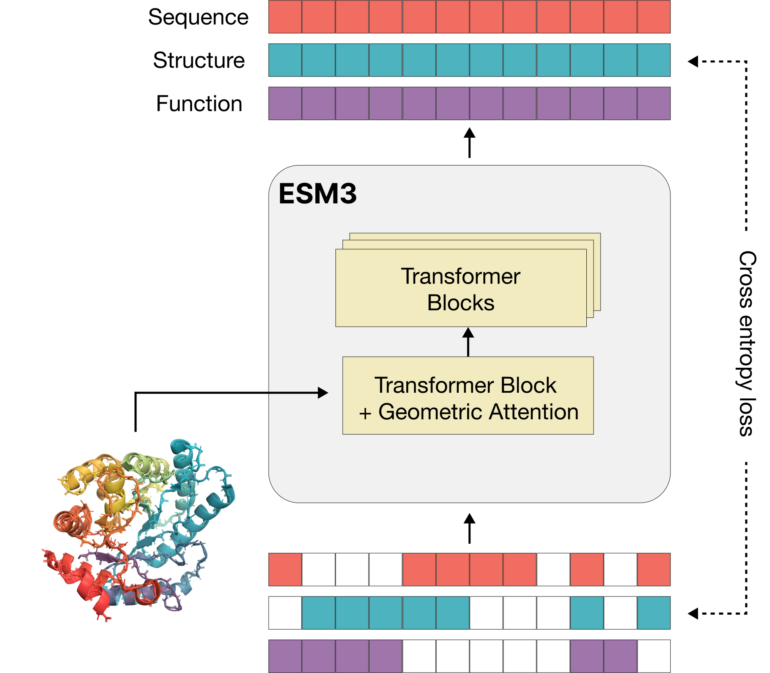
Das Ergebnis ist ein Modell, das Proteine in einem organisierten Raum betrachtet, in dem jedes Protein von allen anderen Proteinen umgeben ist, die eine Mutation entfernt sind. "ESM3 arbeitet nicht innerhalb der physikalischen Grenzen der Evolution, sondern konstruiert implizit ein Modell der vielen möglichen Evolutionspfade", erklären die Autoren. Die Evolutionspfade verbinden alle Proteine, ohne dass das übergeordnete System seine Funktion verliert.
ESM3 überspringt 500 Millionen Jahre Evolution
Durch geschickte Prompts kann ESM3 völlig neue funktionelle Proteine erzeugen. Die Wissenschaftler demonstrieren dies am Beispiel des grün fluoreszierenden Proteins (GFP). Sie gaben ESM3 die Sequenz und Struktur einiger Schlüsselreste vor, die für die Fluoreszenz entscheidend sind. Auf dieser Grundlage generierte das Modell schrittweise die restliche Proteinsequenz und -struktur.
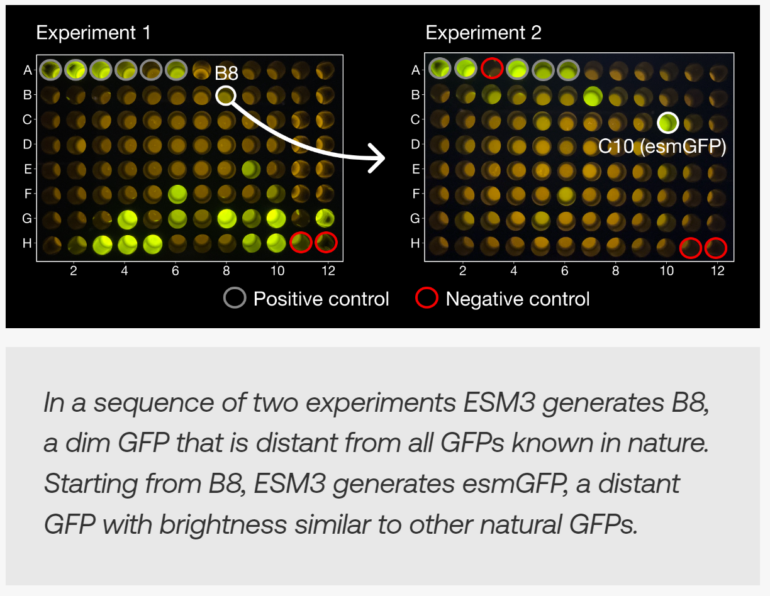
Eines der erzeugten Proteine, "esmGFP", weist eine hohe Leuchtkraft auf, obwohl es sich in 58 % seiner Aminosäuresequenz vom nächsten bekannten fluoreszierenden Protein unterscheidet. Eine so große Veränderung hätte in der Natur mehr als 500 Millionen Jahre gedauert, schätzen die Autoren.
EvolutionaryScale wurde von ehemaligen Meta-Forschern gegründet
Die Studie zeigt erneut das Potenzial von Transformern, die biologische Komplexität von Proteinen zu erfassen und neue Funktionen zu generieren. Das haben die Firmengründer bereits zuvor bewiesen: Sie sind ehemalige Mitglieder der Meta-FAIR Proteingruppe und waren unter anderem an ESMFold beteiligt. Meta hat die Abteilung im August 2023 aufgelöst, während die Konkurrenz von Google mit Deepminds AlphaFold 3 weiter auf diesem Gebiet arbeitet.
Laut dem Team von EvolutionaryScale eröffnet ESM3 nun einen programmgesteuerten Ansatz für das Design von Proteinen, der die Lücke zwischen menschlichen Vorgaben und der Komplexität der Biologie überbrückt. In Zukunft könnte diese Technik zahlreiche Anwendungen in Biotechnologie und Medizin ermöglichen.
Die Forscher betonen aber auch die Notwendigkeit eines verantwortungsvollen Umgangs mit solch leistungsfähigen KI-Modellen. Sie veröffentlichen daher auch eine für Forscher offene Version ESM3-open. Nach Angaben des Teams wurde das Modell von Experten auf seine Sicherheit geprüft. Nach Ansicht der Experten überwiegen die positiven Auswirkungen der Veröffentlichung deutlich die Risiken.
Die vollständigen ESM3-Modelle sollen über eine API mit freiem Zugang für die akademische Forschung zur Verfügung stehen.
KI-News ohne Hype – von Menschen kuratiert
Mit dem THE‑DECODER‑Abo liest du werbefrei und wirst Teil unserer Community: Diskutiere im Kommentarsystem, erhalte unseren wöchentlichen KI‑Newsletter, 6× im Jahr den „KI Radar"‑Frontier‑Newsletter mit den neuesten Entwicklungen aus der Spitze der KI‑Forschung, bis zu 25 % Rabatt auf KI Pro‑Events und Zugriff auf das komplette Archiv der letzten zehn Jahre.
Jetzt abonnierenKI-News ohne Hype
Von Menschen kuratiert.
- Mehr als 20 Prozent Launch-Rabatt.
- Lesen ohne Ablenkung – keine Google-Werbebanner.
- Zugang zum Kommentarsystem und Austausch mit der Community.
- Wöchentlicher KI-Newsletter.
- 6× jährlich: „KI Radar“ – Deep-Dives zu den wichtigsten KI-Themen.
- Bis zu 25 % Rabatt auf KI Pro Online-Events.
- Zugang zum kompletten Archiv der letzten zehn Jahre.
- Die neuesten KI‑Infos von The Decoder – klar und auf den Punkt.