In einer neuen Studie zeigen Deepmind und Kollegen von Isomorphic Labs erste Ergebnisse einer neuen Version von AlphaFold, die die vollautomatische Strukturvorhersage selbst komplexer Moleküle einen Schritt näher bringt.
Das Team von Google Deepmind AlphaFold und Isomorphic Labs hat heute das neueste AlphaFold-Modell vorgestellt. Nach Angaben der Unternehmen kann das aktualisierte Modell nun die Struktur fast jedes Moleküls in der Protein Data Bank (PDB) vorhersagen, oft mit atomarer Genauigkeit. Diese Entwicklung sei ein wichtiger Schritt auf dem Weg zu einem besseren Verständnis der komplexen biologischen Mechanismen innerhalb von Zellen.
Seit seiner Einführung im Jahr 2020 hat AlphaFold die Proteinstrukturvorhersage weltweit beeinflusst. Die neueste Version des Modells geht über Proteine hinaus und umfasst ein breites Spektrum biologisch relevanter Moleküle wie Liganden, Nukleinsäuren und posttranslationale Modifikationen. Diese Strukturen sind für das Verständnis biologischer Mechanismen in der Zelle von entscheidender Bedeutung und waren bisher laut Deepmind nur schwer mit hoher Genauigkeit vorherzusagen.
Deepminds AlphaFold übertrifft spezialisierte Vohersagemodelle
Bei der Vorhersage von Protein-Ligand-Wechselwirkungen übertrifft AlphaFold herkömmliche Methoden um etwa 20 Prozent und kann auch Vorhersagen für völlig neue Proteine treffen, die bisher noch nicht strukturell charakterisiert wurden. Auch bei Protein-Protein-Kontakten konnte die Genauigkeit im Vergleich zu früheren Versionen zum Teil erheblich gesteigert werden, insbesondere in bestimmten Kategorien wie Antikörper-Bindungsstrukturen.
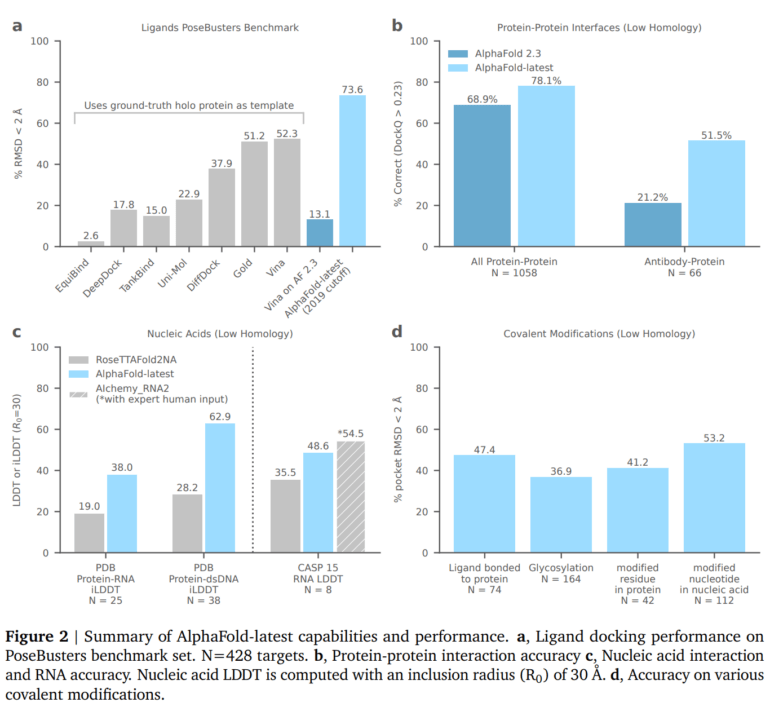
Bei den Protein-Nukleinsäure-Schnittstellen übertrifft die neue Version von AlphaFold konkurrierende Systeme wie RoseTTAFold2NA, während es bei der Vorhersage von RNA-Strukturen zwar andere automatisierte Methoden übertrifft, aber leicht hinter dem besten CASP15-Teilnehmer liegt, der manuelle Eingriffe durch menschliche Experten verwendet. Das verbesserte AlphaFold-Modell könne daher erhebliche Auswirkungen auf die Entdeckung von Medikamenten haben, bei der die Vorhersage relevanter Strukturen wie Antikörperbindungen und Protein-Ligand-Bindungen eine wichtige Rolle spielt.
Neue Version von AlphaFold bringt vollautomatische Strukturvorhersage näher
Obwohl sich das vorgestellte Modell noch in der aktiven Entwicklung befindet, ist das Team der Ansicht, dass es bereits jetzt zeigt, dass die computergestützte Strukturvorhersage von allgemeinen Biomolekülen durch maschinelle Lernmodelle wie AlphaFold möglich ist und viele aufregende zukünftige Forschungswege zum Verständnis der Biologie eröffnet.
Dem Ziel einer vollautomatischen Strukturvorhersage direkt aus der Sequenz scheint das Unternehmen damit einen Schritt näher gekommen zu sein.