Metas ESMFold rechnet 60-mal schneller als AlphaFold
Meta AI veröffentlicht eine Protein-Datenbank mit der Struktur hunderter Millionen Proteinen. Möglich ist das durch ein neues KI-Modell, das deutlich schneller rechnet als Deepminds AlphaFold.
Im Dezember 2020 zeigte Deepmind AlphaFold 2, ein KI-System für die Vorhersage von Proteinfaltungen. Das KI-System ist so viel schneller als alternative Methoden, dass es von manchen Wissenschaftler:innen als eine Lösung für das knapp 50 Jahre alte Problem der Proteinfaltung gesehen wird. Im Juli 2021 veröffentlichte Deepmind dann AlphaFold 2 als Open-Source und eine umfassende Protein-Datenbank.
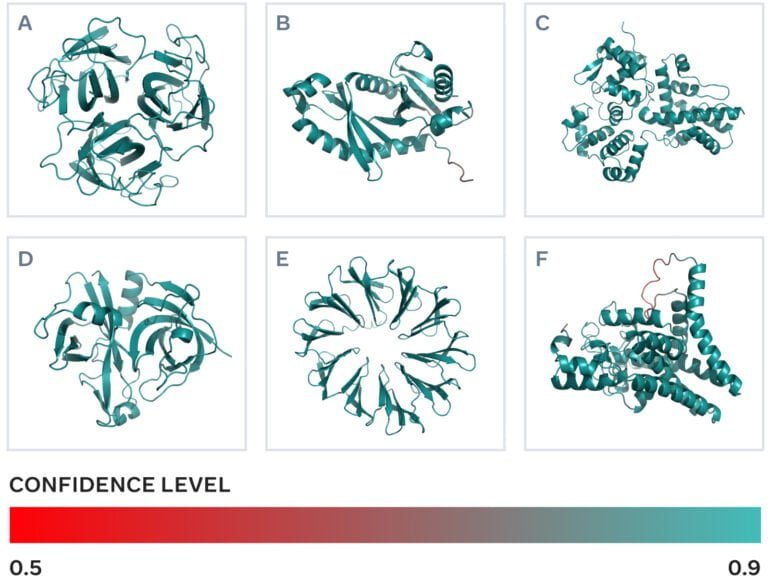
Nun zeigen Forschende von Meta ebenfalls ihre Fortschritte beim Einsatz von KI-Modellen für die Vorhersage von Proteinstrukturen.
ESM Metagenomic Altas Datenbank umfasst hunderte Millionen Strukturvorhersagen
Der von Meta veröffentlichte ESM Metagenomic Atlas umfasst Strukturvorhersagen für 617 Millionen Proteine, die in Mikroben im Boden, im Meer oder im menschlichen Körper vorkommen. Die Anzahl solcher Proteine übertrifft bei Weitem jene aus tierischem und pflanzlichem Leben. Dennoch gehören sie zu den am wenigsten verstandenen Proteinen.
"Das sind die Strukturen, über die wir am wenigsten wissen. Das sind unglaublich mysteriöse Proteine. Ich denke, sie bieten das Potenzial für großartige Einblicke in die Biologie", so Alexander Rives, der Forschungsleiter des Protein-Teams von Meta AI.
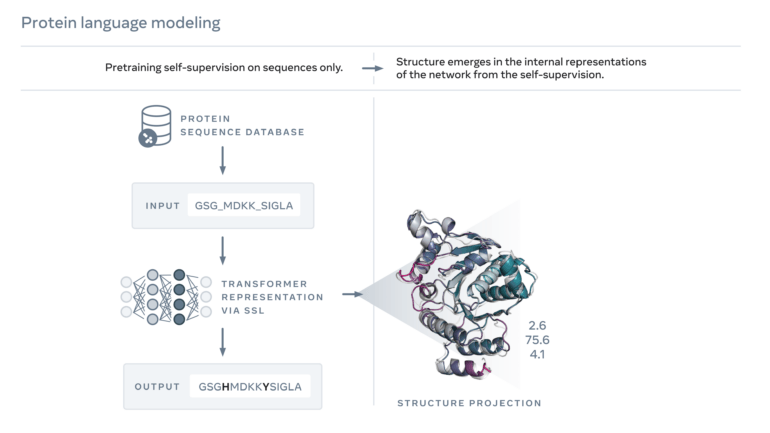
Die Strukturvorhersagen stammen von Metas ESMFold, einem KI-Modell, das im Kern auf ein großes Sprachmodell setzt, das mit Aminosäure-Sequenzen bekannter Proteine trainiert wurde. ESMFold kann nach dem Training Sequenzen vervollständigen und aus diesen im zweiten Schritt Strukturen vorhersagen.
Metas ESMFold ist ungenauer als AlphaFold, doch deutlich schneller
Laut Meta erreicht ESMFold nicht die Genauigkeit von Deepminds AlphaFold in der Strukturvorhersage, kann jedoch Strukturen 60-mal schneller generieren. Damit lässt sich Metas Ansatz deutlich einfacher auf große Datenbanken skalieren, wie im Fall der nun veröffentlichten Datenbank metagenomischer DNA. Die überwiegende Anzahl der Einträge in der Datenbank stammen von Organismen, die noch nie im Labor untersucht wurden.
Announcing the ESM Metagenomic Atlas — the first comprehensive view of the ‘dark matter’ of the protein universe. Made possible by ESMFold, a new breakthrough model for protein folding from Meta AI.
More in our new blog ➡️ https://t.co/LsUhSjzqCf
1/3 pic.twitter.com/5lq48rPv5A
— Meta AI (@MetaAI) November 1, 2022
Für die 617 Millionen Vorhersagen benötigte Metas ESMFold zwei Wochen. Das Modell stufte ein Drittel der Vorhersagen als qualitativ hochwertig ein. Forschende können in diesen Fällen davon ausgehen, dass die Proteinform korrekt ist und in einigen Fällen feinere Details auf atomarer Ebene erkennbar sind.
Meta veröffentlicht neben dem ESM Metagenomic Atlas auch die ESMFold-Modelle sowie ein Pre-Print Paper.
KI-News ohne Hype – von Menschen kuratiert
Mit dem THE‑DECODER‑Abo liest du werbefrei und wirst Teil unserer Community: Diskutiere im Kommentarsystem, erhalte unseren wöchentlichen KI‑Newsletter, 6× im Jahr den „KI Radar"‑Frontier‑Newsletter mit den neuesten Entwicklungen aus der Spitze der KI‑Forschung, bis zu 25 % Rabatt auf KI Pro‑Events und Zugriff auf das komplette Archiv der letzten zehn Jahre.
Jetzt abonnierenKI-News ohne Hype
Von Menschen kuratiert.
- Mehr als 20 Prozent Launch-Rabatt.
- Lesen ohne Ablenkung – keine Google-Werbebanner.
- Zugang zum Kommentarsystem und Austausch mit der Community.
- Wöchentlicher KI-Newsletter.
- 6× jährlich: „KI Radar“ – Deep-Dives zu den wichtigsten KI-Themen.
- Bis zu 25 % Rabatt auf KI Pro Online-Events.
- Zugang zum kompletten Archiv der letzten zehn Jahre.
- Die neuesten KI‑Infos von The Decoder – klar und auf den Punkt.