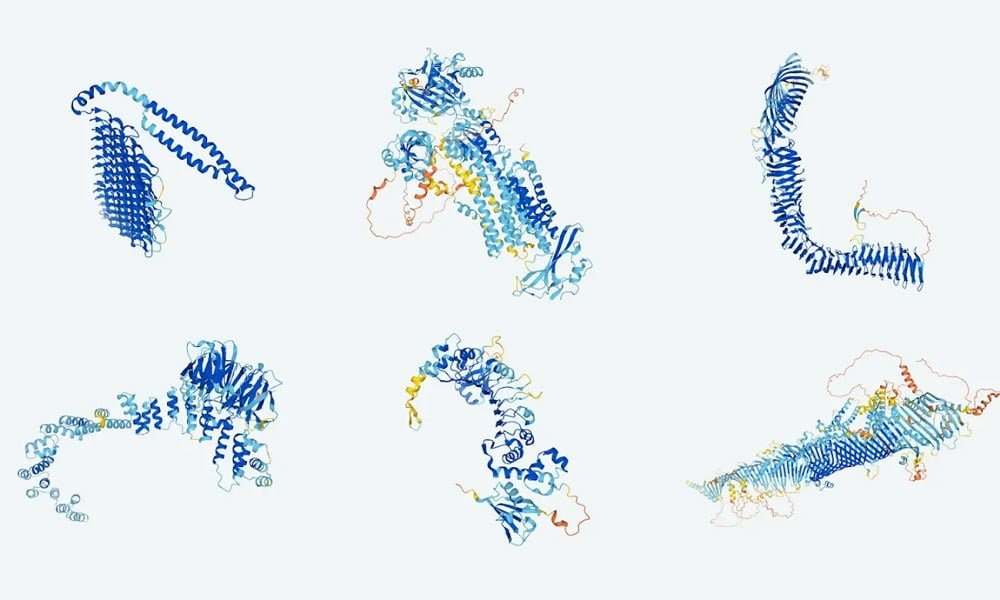
Die Londoner KI-Firma Deepmind stellt der wissenschaftlichen Gemeinschaft die bislang umfangreichste Datenbank zu menschlichen Proteinstrukturen und darüber hinaus zur Verfügung. Sie könnte zur Grundlage für viele wissenschaftliche Durchbrüche in den nächsten Jahren werden.
"Davon habe ich mein Leben lang geträumt, das ist der Grund, weshalb Deepmind gegründet wurde", sagt Demis Hassabis. Die Begeisterung des Deepmind-Gründers entfacht sich an der neuesten Bekanntgabe seines Unternehmens: Deepmind veröffentlicht die "Alphafold Protein Structure Database", die bislang umfangreichste Sammlung prognostizierter Proteinstrukturen.
Um die Dimension der Datenbank zu veranschaulichen: Bislang waren rund 180.000 experimentelle Proteinstrukturen in der Open-Access-Datenbank Protein Data Bank frei für die Wissenschaft zugänglich. Dieser Sammlung fügt Deepmind jetzt circa 350.000 weitere 3D-Strukturen von 20 unterschiedlichen Organismen hinzu. Rund 20.000 der prognostizierten Strukturen gehören zum Menschen, was circa 98 Prozent der Proteinstrukturen im menschlichen Körper entspricht.
Für Deepmind ist die heutige Bekanntgabe erst der Anfang: Das Unternehmen verspricht, dass die Proteinstruktur-Sammlung in den kommenden Monaten deutlich wachsen und "einen großen Teil" aller 100 Millionen Proteine abdecken soll, die in der UniRef90-Datenbank katalogisiert sind. Das entspricht laut Deepmind "fast aller sequenzierten Proteine, die der Wissenschaft bekannt sind."
"Wir glauben, dass diese Arbeit den bedeutendsten Beitrag darstellt, den KI bisher zum Fortschritt des wissenschaftlichen Wissens geleistet hat, und sie ist ein großartiges Beispiel für den Nutzen, den KI der Gesellschaft bringen kann", sagt Hassabis.
Deepminds Protein-Datenbank: Nährboden für wissenschaftliche Durchbrüche in vielen Bereichen
Deepmind veröffentlicht die Datenbank gemeinsam mit dem Europäischen Institut für Bioinformatik des Europäischen Laboratoriums für Molekularbiologie (EMBL-EBI). Die Organisationen hoffen, dass die Informationen Forschern dabei helfen, etwa Wirkstoffe gegen Krankheiten zu entdecken oder neue Nahrungsmittel zu entwickeln.
"Die Anwendungen sind nur durch unsere Vorstellungskraft begrenzt - aber auf einer grundlegenderen Ebene wird die Alphafold-Datenbank unser Verständnis der Funktionsweise von Proteinen und ihrer Rolle in den grundlegenden Prozessen des Lebens verbessern", sagt Prof. Edith Heard, Generaldirektorin des EMBL.
Elizabeth Blackburn, Nobelpreisträgerin 2009 für Physiologie oder Medizin, geht davon aus, dass Deepminds Veröffentlichung der wissenschaftlichen Gemeinschaft "neue Einblicke in die biologische Bedeutung der Genomsequenz eröffnen" wird.
Alle Prognosen in der Datenbank wurden mit Deepminds KI-System Alphafold erstellt, das seit Mitte Juli als Open-Source-Version verfügbar ist. Die Vorhersagen von Alphafold 2.0 erreichten zuvor in Testszenarien eine viel höhere Genauigkeit in deutlicher kürzerer Zeit als bisherige Methoden zur Proteinstrukturprognose.
"Unser Traum ist, dass AlphaFold durch die Bereitstellung dieses grundlegenden Wissens unzähligen weiteren Wissenschaftlern bei ihrer Arbeit hilft und völlig neue Ansätze für wissenschaftliche Entdeckungen eröffnet", sagt Hasabis.
Die Informationen in der Datenbank sind für die akademische und kommerzielle Forschung kostenlos und ohne Einschränkungen nutzbar. Deepminds Datenbank ist hier online verfügbar.
Quellen: Pressemitteilung, Deepmind