Nvidia, Evozyne, InstaDeep und Forschende der TU München zeigen auf der JP Morgan Healthcare Konferenz neue Fortschritte im Einsatz von KI in der Biologie.
Fortschritte in generativen KI-Modellen für Sprache und Bilder verändern den Markt für die maschinelle Verarbeitung natürlicher Sprache (NLP), Kunst und Design. Doch die zugrundeliegenden Technologien wie Transformer, Diffusion-Modelle oder Variational Autoencoders (VAE) und Methoden, wie unüberwachtes Lernen mit gigantischen Datenmengen, beweisen sich auch außerhalb dieser Domänen.
Ein erfolgversprechendes Anwendungsgebiet ist die Bioinformatik, in der Modelle wie Deepminds AlphaFold 2 oder Metas ESMFold die Struktur von Proteinen vorhersagen oder der Siegeszug der Diffusions-Modelle wie Stable Diffusion eine neue Ära im Protein-Design einläuten soll.
Allein 2022 wurden knapp 1.000 wissenschaftliche Arbeiten auf Arxiv zum Einsatz von KI in der Biologie veröffentlicht. Bis 2025 könnten mehr als 30 Prozent der neuen Medikamente und Materialien systematisch mit Hilfe generativer KI-Techniken entdeckt werden, heißt es etwa im Gartner-Report "Innovation Insight for Generative AI".
Nvidia kooperiert mit Start-ups und Forschenden für Fortschritte in der Bioinformatik
Auf der diesjährigen JP Morgan Healthcare Konferenz stellt Nvidia nun die Ergebnisse zweier Kooperation mit Start-ups und Forschenden vor: das Genomik-Sprachmodell Nucleotide Transformer und das generative Protein-Modell ProT-VAE.
Der Nucleotide Transformer entstand in einer Kollaboration zwischen dem kürzlich von BionTech übernommenen InstaDeep, der Technischen Universität München und Nvidia. Das Team trainierte verschiedene Modellgrößen mit Daten von bis zu 174 Milliarden Nukleotiden verschiedener Spezies auf Nvidias Cambridge-1 Supercomputer und folgt damit dem Erfolgsrezept großer Sprachmodelle wie GPT-3: große Modelle, gigantischer Datensatz und viel Rechenleistung.
Wie erhofft, stieg die Leistung des Nucleotide Transformer mit Modellgröße und Datenmenge. Das Team testete das Modell in 19 Benchmarks und erreichte in 15 eine gleichwertige oder bessere Leistung als andere Modelle, die speziell für diese Aufgaben trainiert wurden. Der Transformer soll in Zukunft etwa helfen, DNA-Sequenzen in RNA und Proteine zu übersetzen.
"Wir glauben, dass dies die ersten Ergebnisse sind, die eindeutig die Machbarkeit der Entwicklung von Foundation Models in der Genomik zeigen, die wirklich über mehrere Aufgaben hinweg verallgemeinert werden können", so Karim Beguir, CEO von InstaDeep. "In vielerlei Hinsicht spiegeln diese Ergebnisse wider, was wir in den letzten Jahren bei der Entwicklung anpassungsfähiger Foundation Models in der Verarbeitung natürlicher Sprache gesehen haben, und es ist unglaublich spannend zu sehen, dass dies nun auf solch herausfordernde Probleme in der Arzneimittelforschung und der menschlichen Gesundheit angewendet wird."
KI-Modell ProT-VAE generiert neue Proteine
Forschende des Start-ups Evozyne gehen einen Schritt weiter: Sie nutzten Nvidias BioNeMo-Plattform, um mit dem generativen Modell ProT-VAE neue Proteine zu generieren. Modelle wie AlphaFold oder ESMFold sagen aus Protein-Sequenzen deren Struktur vorher, ProT-VAE soll dagegen aus den Sequenzen direkt Funktionen ableiten und so gezielt neue Proteine generieren, die eine bestimmte Funktion übernehmen.
Die Fähigkeit, Proteine mit vorher festgelegten Funktionen zu entwickeln, ist ein zentrales Ziel der synthetischen Biologie und hat Potenzial, etwa die Medizin, die Bioverfahrenstechnik oder den Energiesektor zu revolutionieren.
Das Problem: Allein mit den natürlich vorkommenden Aminosäuren gibt es deutlich mehr mögliche Proteine als Protonen im sichtbaren Universum.
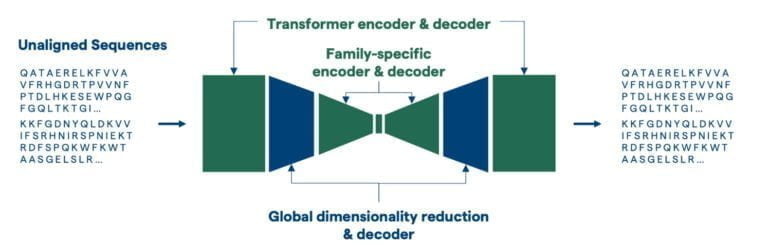
Die Lösung sieht Evozyne im "Machine Learning Guided Protein Engineering" mit ProT-VAE. Das Modell legt ein VAE-Netz zwischen einen von Nvidia vortrainierten Protein-Transformer-Encoder und -Decoder. Das VAE-Netz wird anschließend für eine bestimmte Protein-Familie trainiert, in der neue Proteine generiert werden sollen. In dem generativen Prozess kann das Modell jedoch weiter von den umfassenden Repräsentationen des ProtT5-Transformers profitieren, der während Nvidias Training Sequenzen von Aminosäuren in Millionen von Proteinen verarbeitet hat.
Um das Modell zu testen, entwickelte das Team unter anderem eine Variante des menschlichen PAH-Proteins. Mutationen am PAH-Gen können dessen Aktivität einschränken und zu Stoffwechselerkrankungen führen, die etwa die geistige Entwicklung stört und zu Epilepsie führen kann. Laut den Forschenden entwarf ProT-VAE eine Variante mit 51 Mutationen, 85 Prozent Sequenz-Übereinstimmung und einer um den Faktor 2,5 verbesserten Funktion.
Wir gehen davon aus, dass das Modell eine erweiterbare und generische Plattform für maschinelles Lernen und gezielte Evolutionskampagnen für das datengesteuerte Design neuartiger synthetischer Proteine mit "übernatürlichen" Funktionen bieten kann.
Aus dem Paper
Dieser Prozess habe bis vor kurzem Monate bis Jahre in Anspruch genommen. Mit ProT-VAE ließe sich diese Zeit auf wenige Wochen reduzieren.